1.获取IGV软件
2.运行软件
3.加载基因组4.加载注释文件5.导入样品比对文件6.调整图像7.导出图像
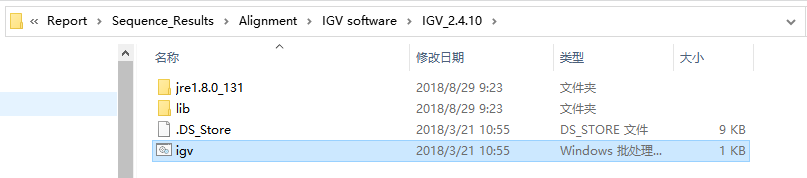
云序生物测序报告中:Report>Sequence_Results>Alignment>IGV software中已经提供了IGV_2.4.10版本的软件压缩包,解压后打开可直接点击igv(.bat)文件运行,无需安装。
*如果运行报错或操作界面错乱,可能由于java版本不对应,可尝试方式二下载java included IGV。
自行从网站下载:
http://software.broadinstitute.org/software/igv/download
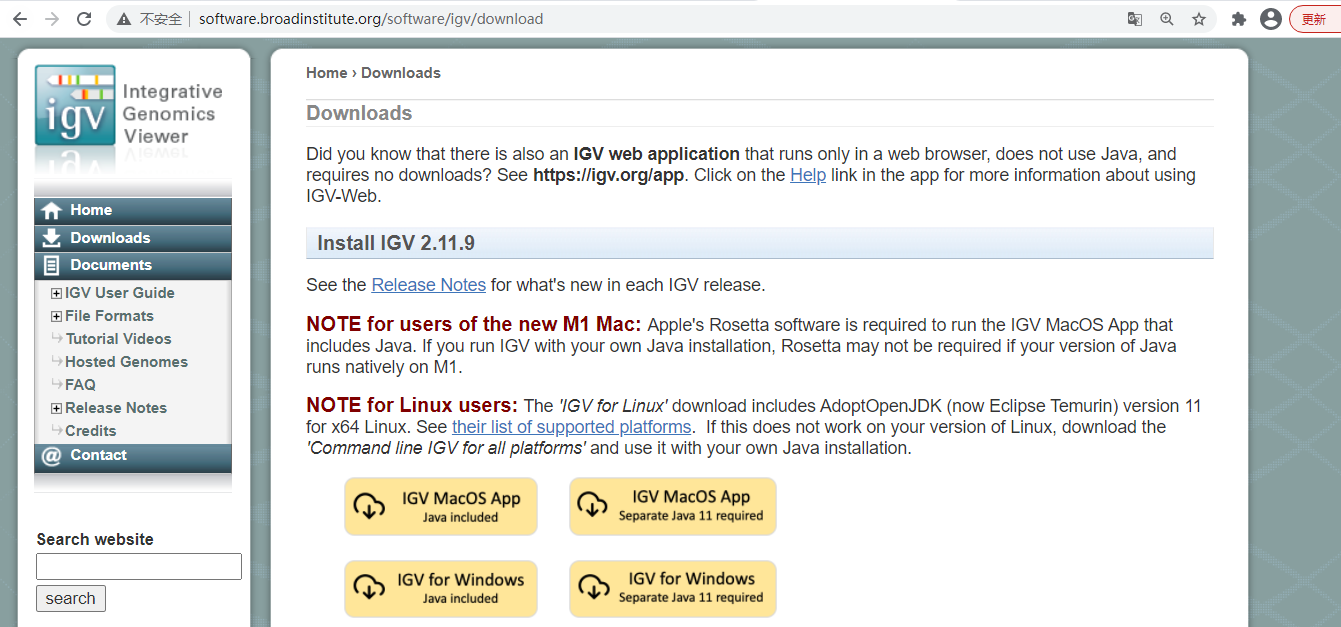
建议选java included的版本。
下载的版本需要安装:
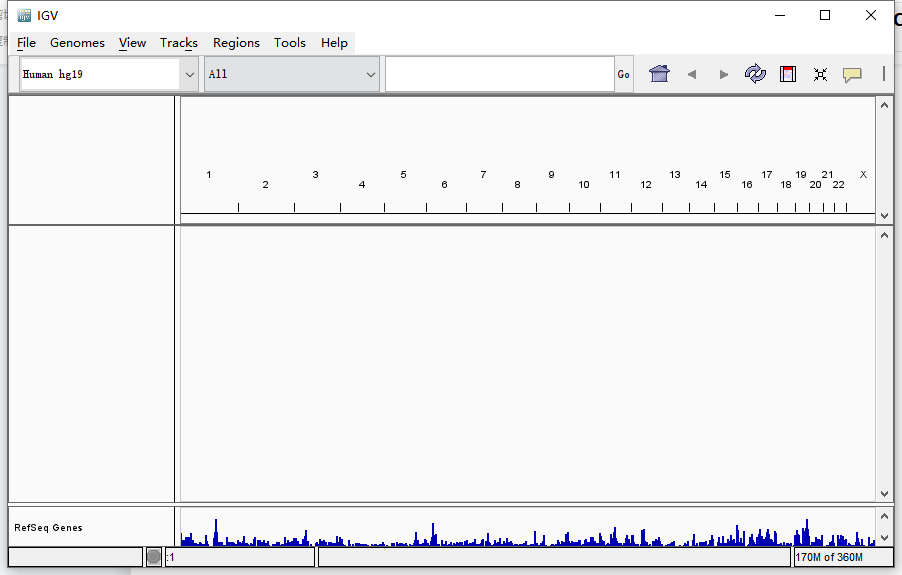
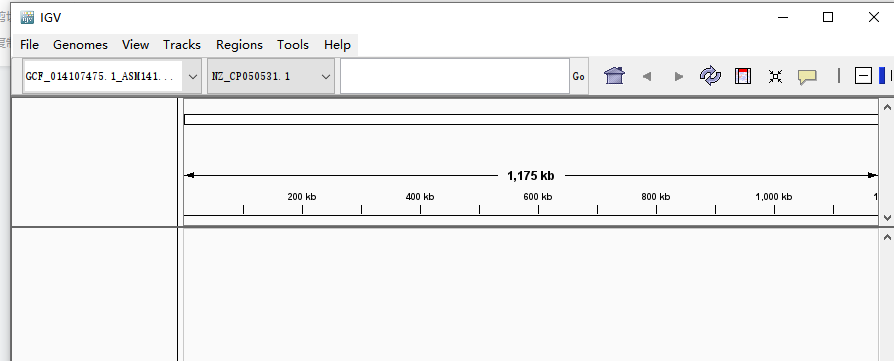
加载完毕Loading Genome...框消失,操作界面如下
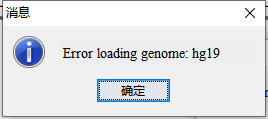
*如果出现如下报错,原因可能是网络信号不佳,或IGV服务系连接问题。如果确认网络状态正常仍不能解决,可改其他时间尝试,或自行手动导入自己储存在本地的基因组(见下文3.加载基因组)。
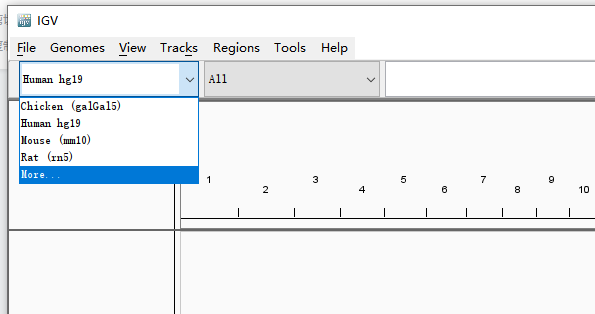
左上角可看到基因组信息,一般软件初始会默认加载的人类基因组Human hg19版本。如果测序数据分析使用的是其他基因组,需要在此选择,此处包含了一些常见基因组版本可直接选择。点击More有更多的基因组版本可供选择。务必与测序分析所使用的版本保持一致。
(云序生物提供的测序服务,使用的基因组版本见报告中:
Report>Sequence_Results>Supplementary>Sample & groups表格中Genome build)
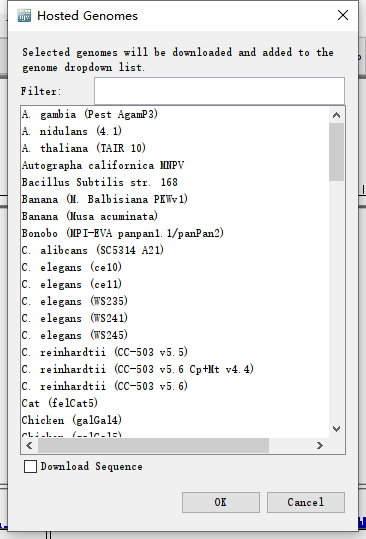
方式二:
如果是特殊物种不在此列的(或网络等原因加载失败的),则需要自行导入基因组:菜单选择Genomes>Load Genome from File...
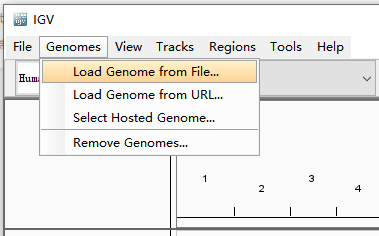
打开自己预先储存在本地的基因组文件。(.fa文件)
(云序生物提供的特殊物种的测序服务,基因组文件如非客户提供,可通过销售提交申请)
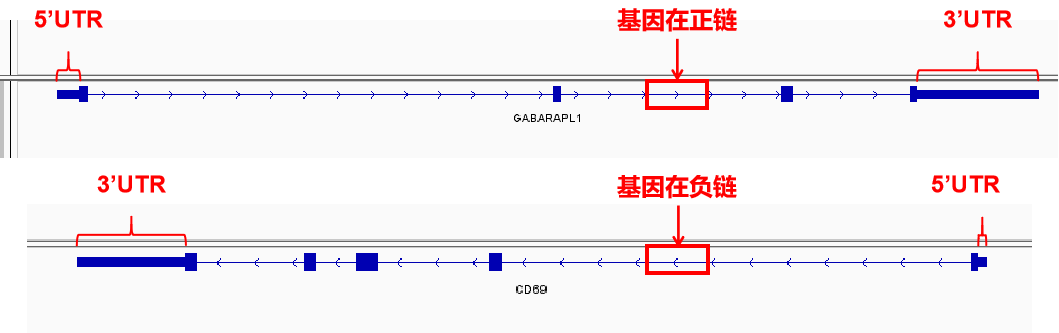
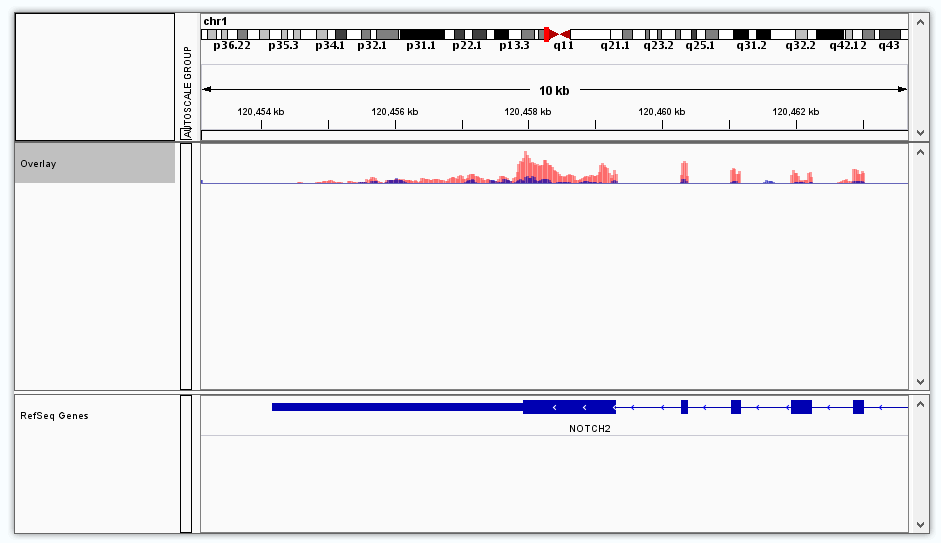
选择染色体并且zoom in之后可以看到基因组上各位置的具体基因和转录本。
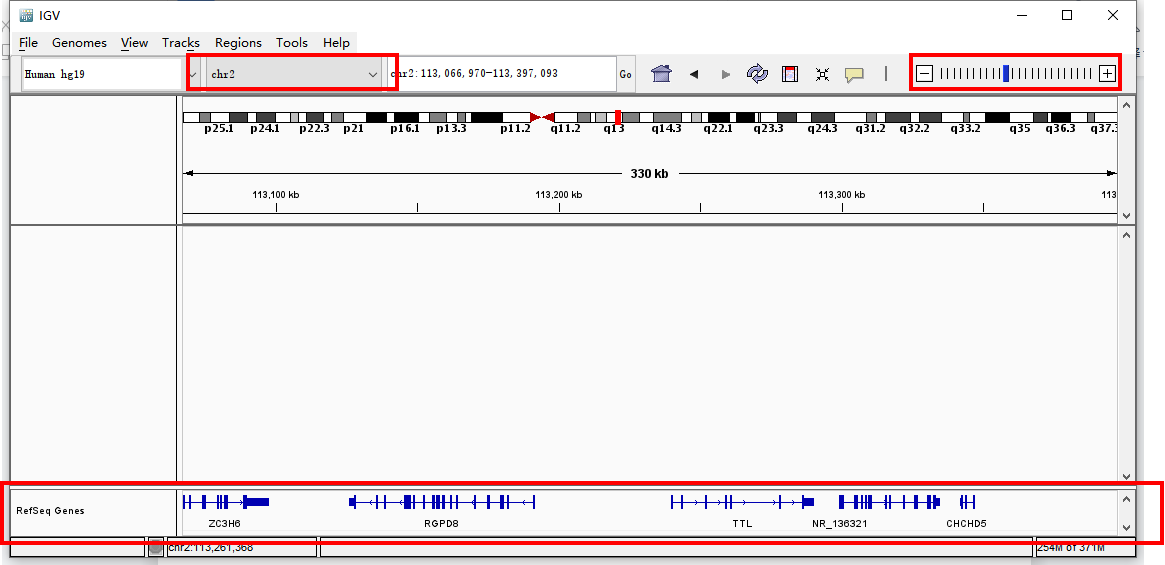
转录本上线性为内含子区域,蓝色矩形为外显子区域,其中两端的较窄的矩形为UTR区。转录本上箭头方向向右->->->-,则该基因在正链上,左侧的UTR区为5’UTR;转录本上箭头方向向左-<-<-<-,则该基因在负链上,右侧的UTR区为5’UTR。
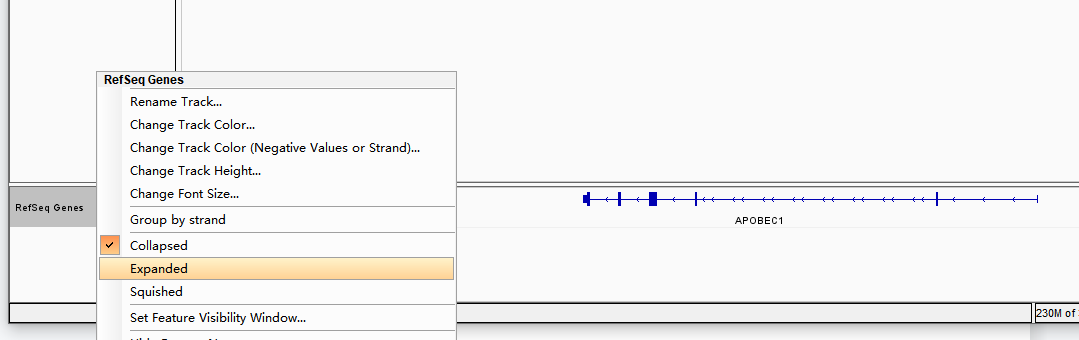
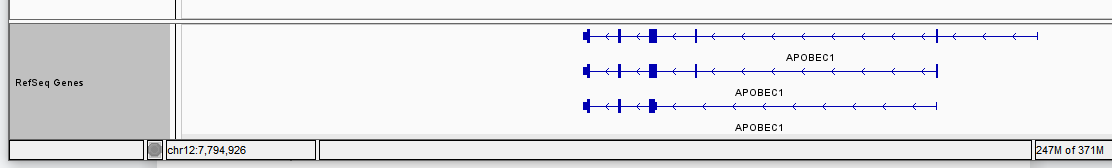
有箭头间隔不均匀的,说明该基因有多个转录本,在此重叠了。可右键点击左侧track名(RefSeq Genes)选择“Expanded”模式查看多个转录本的情况。
有时分析所用的注释文件与IGV默认加载的注释信息有出入,想要对照自己所用的注释信息来查看,可将自己使用的注释文件(.gtf)拖入红框区域。
(云序生物提供的测序服务,使用的注释文件见:Report>Sequence_Results>Alignment)
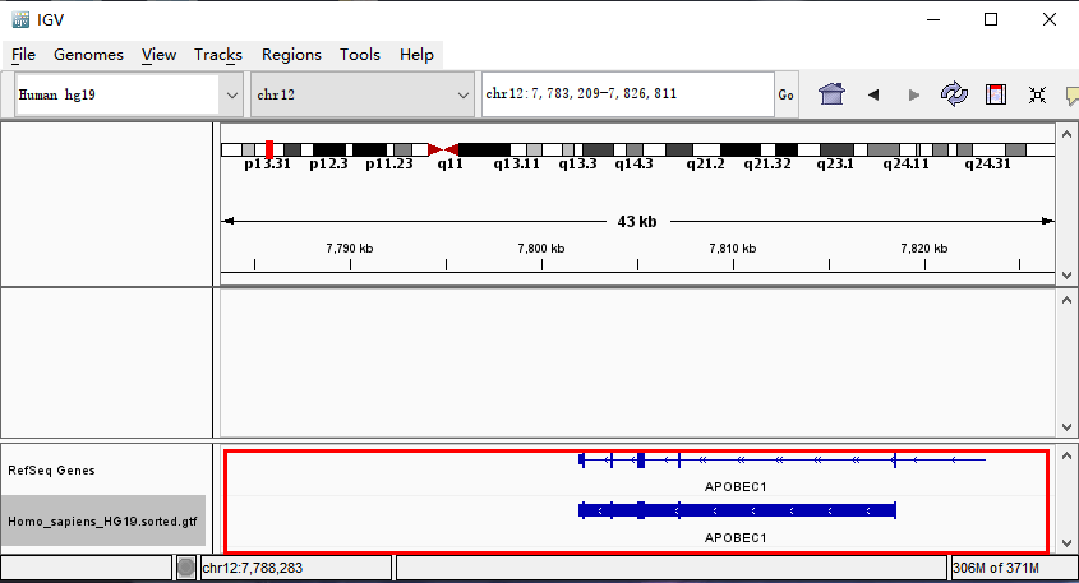
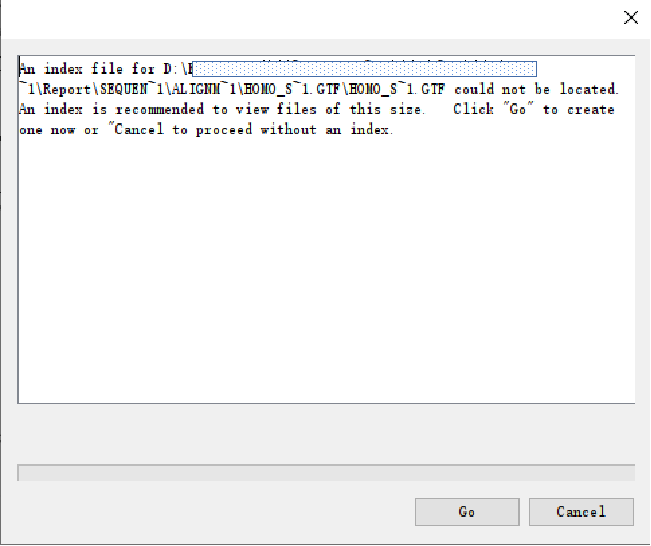
*出现建议建立索引文件的提示,可以点Go等待自动生成索引文件后再次拖动gtf文件进来(推荐),但如果gtf文件是没有sorted过的(文件名中没有.sorted.字样)则无法建立索引文件,如果gtf文件是由云序生物提供的,可联系销售。也可以点cancel忽略继续进行(可能加载会较慢)。

将bam文件拖入红框区域。
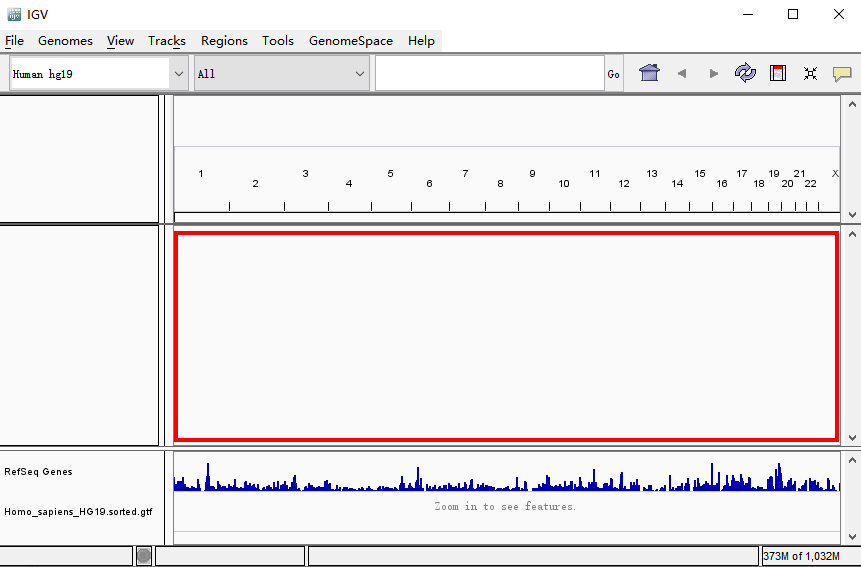
样品刚拖入右侧时,有可能只会显示灰色字“zoom in to see features”。这只是提示需要放大到一定程度才会显示出峰。

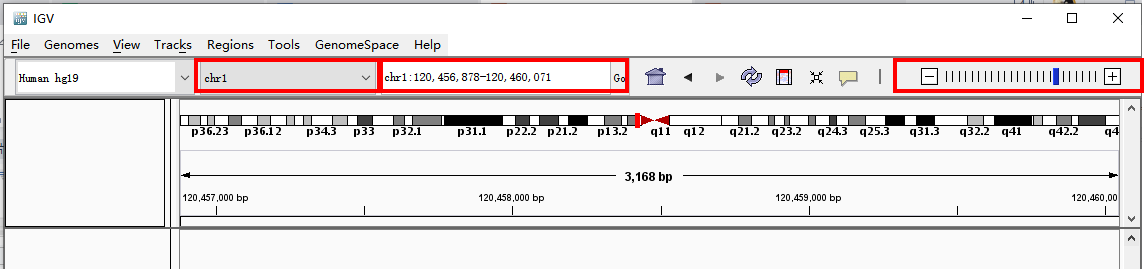
界面上方可以选择要观察的染色体、输入染色体上具体位置或者直接输入基因名,也可以点“-”和“+”来放大和缩小区域。Bam文件在缩放区域时可能会较慢需要耐心等待。
Bam文件加载好后,出现两条track,上面一条coverage即通常可视化图中展示的峰形,下面一条track展现的是每一条read在基因组上的比对情况。
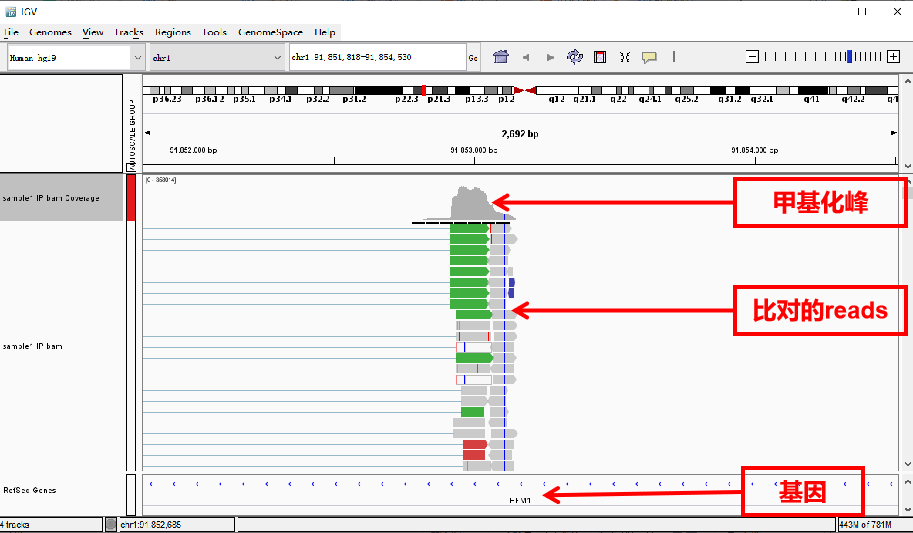
作图时可以右键点击track名选择隐藏这条alignment track,以便让出位置展示多个样品中的甲基化峰。
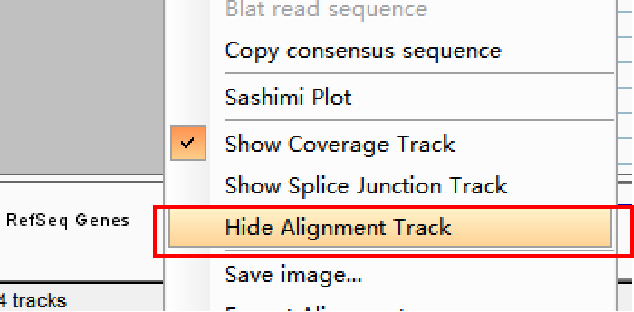
方式二:使用tdf文件进行可视化
由于bam文件较大,读取和调整较慢,可以通过IGV内自带的IGV Tools将bam文件转化为tdf文件来进行可视化。相较bam文件,tdf文件去除了alignment track的信息只保留了coverage信息,但文件变小便于快速操作,并且用tdf文件操作时可以进行多样品叠加展示(见后文6调整图像)。
在菜单选Tools>Run igvtools...
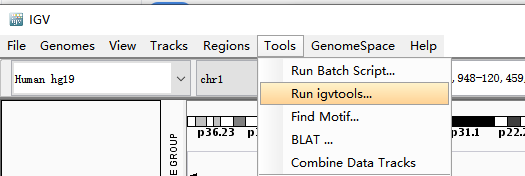
点击Input File后面的browse,选择要转化的bam文件。
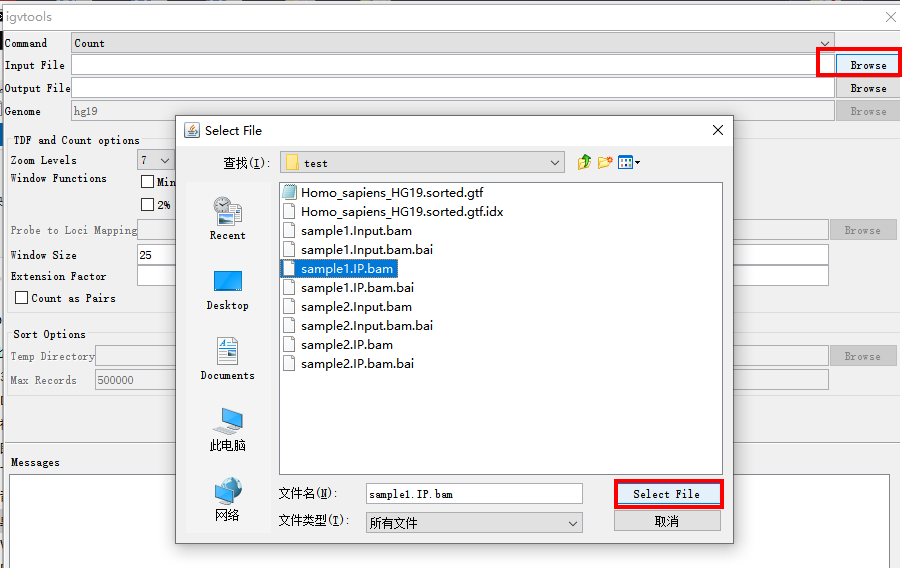
选择好以后,Output File会自动命名,不需要再选择。点击Run进行转化。
*有时出现WARNING: BAM index file...bam.bai is older than ...bam,是提示这个索引文件bai文件比bam文件生成的时间要早,而正常情况下理当先有bam再建立它的bai文件,因此软件提醒注意是否文件有误。一般可能只是由于涉及到原始数据二次下载导致文件生成时间改变,可以忽略这个提示。
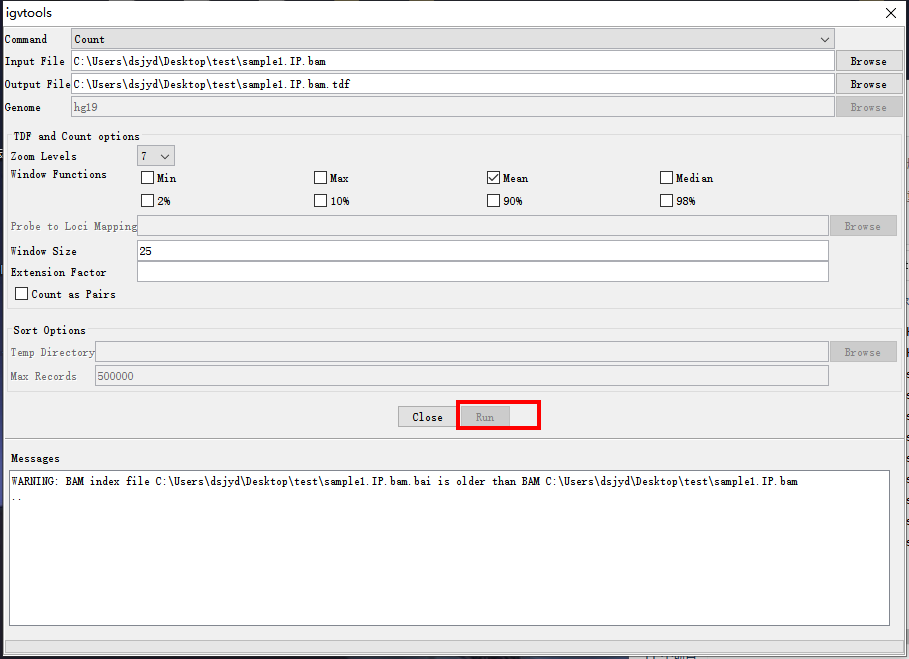
点击Run之后等待其转换,需要一定时间。此时可以看到Run变灰,并且等待一段时间后在Message信息框可以观察到“..”点逐渐增加,说明正在转换中。完成后会显示“100%”和“Done”。
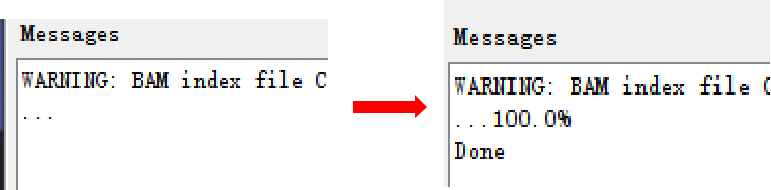
生成的tdf文件被保存在bam同文件夹中。在igvtools中重新选择其他Input File可逐个进行转换。
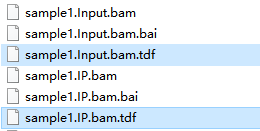
*若tdf文件大小为0或特别小,说明转换未完成或不完整,无法使用,尝试重新进行转换。
tdf文件的使用方式同bam一样,直接将文件拖入IGV。
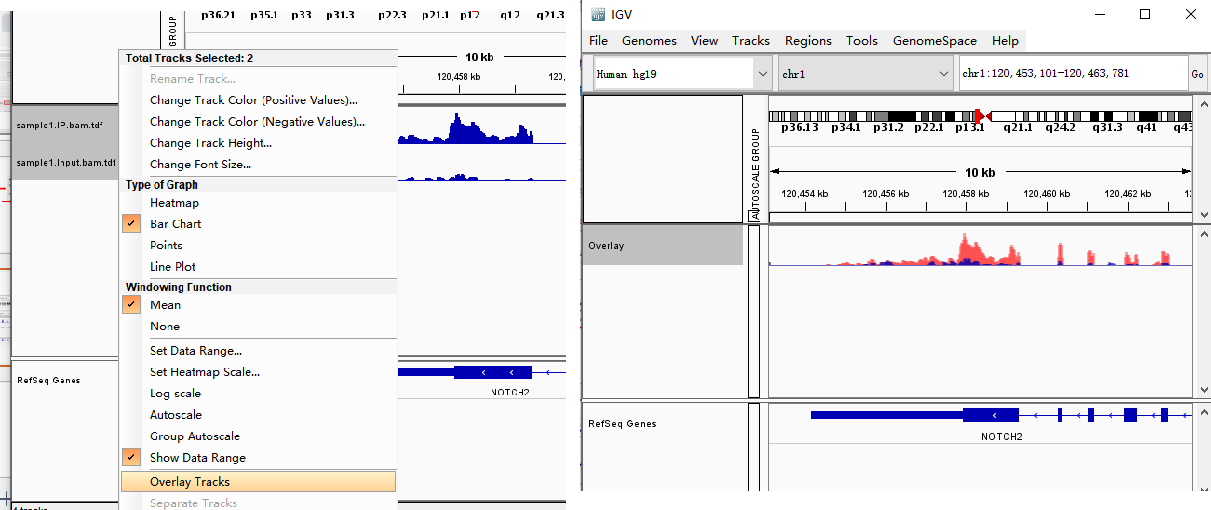
右键样品名可以改样品名(Rename Track)、更改track的高度、颜色、名字字体大小(Change...)等基本操作。
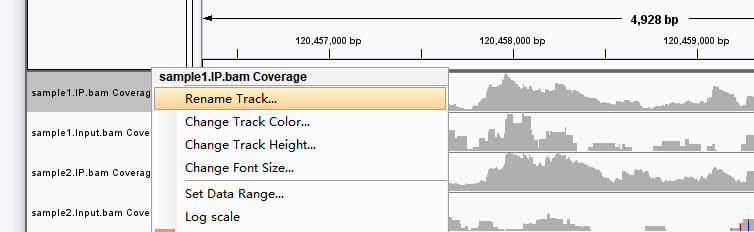
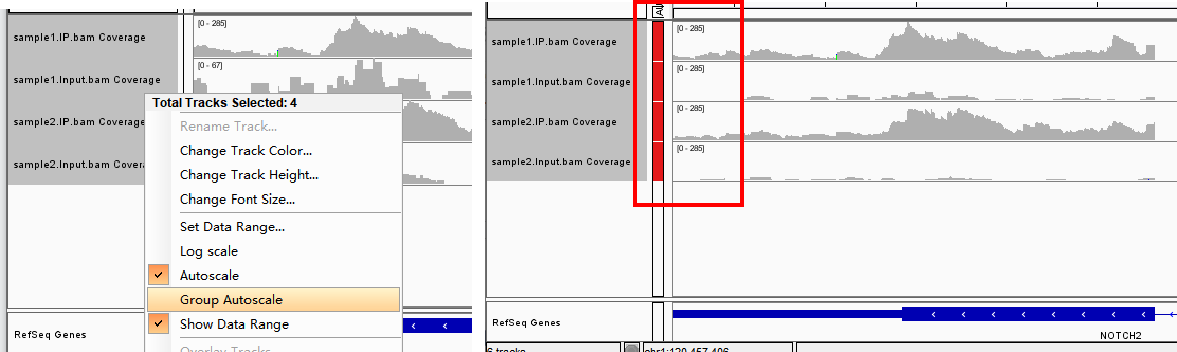
要对比多个样品中甲基化峰时,会需要将各样品纵坐标scale调整至一致才便于直观比较。

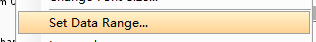
可以 1)根据甲基化峰信号值手动调整个别样品的scale:右键点样本名选Set Data Range后输入最大值;
2)单个样品自动调节至合适的scale:右键点样本名选Autoscale,会自动调整到一个可以显露出该视窗内该样本最高信号值的scale;

3)也可以同时选中要统一的所有样品名(按住shift或者control点选)然后右键选择Group Autoscale,则全部样品会被统一采用一个相同的scale。
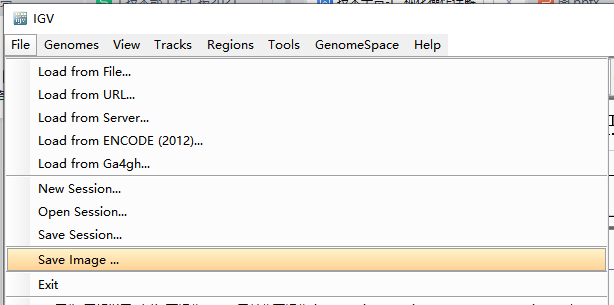
有三种文件格式可以选择。
导出的图片为当前视窗下的全部内容。
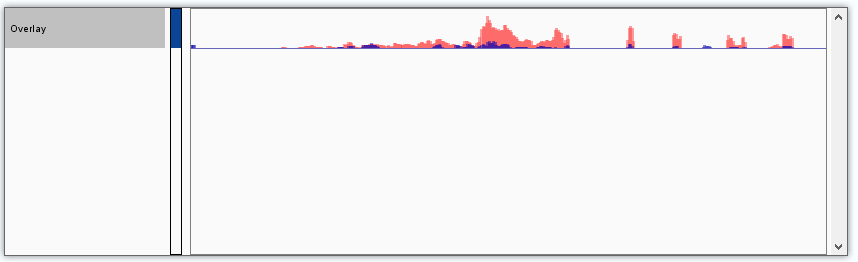
或右键track名,选择Save Image则仅导出样品track的图像。
**有文献IGV可视化图上下方有其他个性化的注释信息、图形,甲基化区域有颜色高亮或框,为IGV外画图软件(PS等)自行添加。
**IGV中其他详细信息,可参阅IGV官网http://www.broadinstitute.org/igv/
云序相关产品
上海云序生物科技有限公司 商家主页
地 址: 上海市松江区莘砖公路518号24号楼4楼
联系人: 戴小姐
电 话: 021-64878766
传 真: 021-64878766
Email:market@cloud-seq.com.cn;liuqingqing@cloud-seq.com.cn
相关咨询
杨宝峰院士团队RNA修饰又一成果 | 云序ac4C acRIP-seq助力揭示心脏I/R损伤的作用机制 (2024-12-03T00:00 浏览数:15631)
杨宝峰院士团队最新成果 | 云序助力揭示RNA修饰m7G调控心肌肥厚的机制研究 (2024-11-13T00:00 浏览数:17053)
Nature子刊| 重磅综述!一文总结「m6A修饰非编码RNAs」在各类肿瘤中的调控机制及作用 (暂无发布时间 浏览数:12855)
研究速览-eccDNA 2023年最新进展大放送! (暂无发布时间 浏览数:15077)
云序生物MeRIP-qPCR技术干货 (暂无发布时间 浏览数:14982)
技术干货| “eccDNA碱基序列的获取及引物设计”方法教程 (暂无发布时间 浏览数:13759)
云序客户m6A高分文章|揭示组蛋白乙酰化与m6A修饰在眼部黑色素瘤发生中的共同作用机制 (暂无发布时间 浏览数:7436)
Nat Biotechnol IF=47 | BID-seq:一种基于单碱基分辨率的假尿嘧啶(Ψ)修饰定量测序检测方法 (暂无发布时间 浏览数:7405)
北大伊成器团队Nature Reviews重磅发文:非m6A热门修饰调控与功能一文速览! (暂无发布时间 浏览数:11655)
用户文章m6A专题|IF=9.8|m6A去甲基化酶ALKBH5缺乏会加重钴致神经退行性损伤 (暂无发布时间 浏览数:7373)
